Cuadrícula de la comunidad mundial - World Community Grid
| Desarrollador (es) | Instituto de Investigación Krembil |
|---|---|
| Versión inicial | 16 de noviembre de 2004 |
| Lanzamiento estable | 7.16.11 |
| Estado del desarrollo | Activo |
| Sistema operativo | Microsoft Windows , Linux , Android , macOS |
| Plataforma | BOINC |
| Escribe | Informática voluntaria |
| Rendimiento medio | 800 TFLOPS |
| Usuarios activos | 33,255 |
| Usuarios totales | 101,949 |
| Anfitriones activos | 154,119 |
| Total de hosts | 183,354 |
| Sitio web | worldcommunitygrid.org |
World Community Grid ( WCG ) es un esfuerzo para crear la red informática pública más grande del mundo para abordar proyectos de investigación científica que benefician a la humanidad. Lanzado el 16 de noviembre de 2004, fue coordinado por IBM con el software cliente actualmente disponible para los sistemas operativos Windows , Linux , macOS y Android . En septiembre de 2021, se anunció que IBM transfirió la propiedad al Instituto de Investigación Krembil .
World Community Grid utiliza la potencia de procesamiento no utilizada de los dispositivos de consumo (PC, portátiles, teléfonos inteligentes Android, etc.) para analizar los datos creados por los grupos de investigación que participan en la red. Los proyectos WCG han analizado datos relacionados con el genoma humano , el microbioma humano , VIH , dengue , distrofia muscular , cáncer , influenza , Ébola , virus Zika , tamizaje virtual , rendimiento de cultivos de arroz , energía limpia , purificación de agua y COVID-19 , entre otros. áreas de investigación.
Actualmente hay 5 proyectos activos y 25 proyectos terminados. Varios de estos proyectos han publicado artículos revisados por pares basados en el análisis de los datos generados por WCG. Estos incluyen un documento del proyecto OpenZika sobre el descubrimiento de un compuesto (FAM 3) que inhibe la proteína NS3 Helicasa del virus Zika, reduciendo así la replicación viral hasta en un 86%; un documento de FightAIDS @ home sobre el descubrimiento de nuevas vulnerabilidades en la proteína de la cápside del VIH-1 que pueden permitir un nuevo objetivo farmacológico; un artículo de FightAIDS @ home sobre nuevas técnicas computacionales de descubrimiento de fármacos para obtener resultados más refinados y precisos.
Historia
En 2003, IBM y otros participantes de la investigación patrocinaron el Proyecto de Red de Investigación de Viruela para acelerar el descubrimiento de una cura para la viruela . El estudio de la viruela utilizó una red informática distribuida masiva para analizar la efectividad de los compuestos contra la viruela. El proyecto permitió a los científicos analizar 35 millones de moléculas de fármacos potenciales contra varias proteínas de la viruela para identificar buenos candidatos para convertirse en tratamientos contra la viruela. En las primeras 72 horas, se devolvieron 100.000 resultados. Al final del proyecto, se habían identificado 44 candidatos fuertes para el tratamiento. Basado en el éxito del estudio sobre la viruela, IBM anunció la creación de World Community Grid el 16 de noviembre de 2004, con el objetivo de crear un entorno técnico donde se pudieran procesar otras investigaciones humanitarias.
World Community Grid inicialmente solo era compatible con Windows, utilizando el software propietario Grid MP de United Devices que impulsó los proyectos de computación distribuida de grid.org . La demanda de soporte para Linux condujo a la adición en noviembre de 2005 de la tecnología de grid de Berkeley Open Infrastructure for Network Computing ( BOINC ) de código abierto que impulsa proyectos como SETI @ home y Climateprediction , y se agregó soporte para Mac OS X y Linux desde la introducción de BOINC. . En 2007, World Community Grid migró de Grid MP a BOINC para todas sus plataformas compatibles.
En septiembre de 2021, IBM anunció que había transferido la propiedad de World Community Grid al Instituto de Investigación Krembil . Todo el proceso de transición se llevará a cabo durante los próximos meses.
Escala del proyecto
Al 21 de septiembre de 2021, World Community Grid tenía más de 33,000 cuentas de usuario activas, con más de 154,000 dispositivos activos. En el transcurso del proyecto, se han donado más de 2,000,000 de años acumulados de tiempo de computación y se han completado más de 6 mil millones de unidades de trabajo.
Operación

El software World Community Grid utiliza el tiempo de computación no utilizado de los dispositivos conectados a Internet para realizar cálculos de investigación. Los usuarios instalan el software del cliente WCG en sus dispositivos. Este software funciona en segundo plano, utilizando recursos del sistema de repuesto para procesar el trabajo para WCG. Cuando se completa un trabajo o una unidad de trabajo , el software del cliente lo envía de vuelta a WCG a través de Internet y descarga una nueva unidad de trabajo. Para garantizar la precisión, los servidores WCG envían varias copias de cada unidad de trabajo. Luego, cuando se reciben los resultados, se recopilan y validan entre sí.
World Community Grid ofrece múltiples proyectos humanitarios bajo un solo paraguas. Los usuarios están incluidos en un subconjunto de proyectos de forma predeterminada, pero pueden optar por no participar en los proyectos que deseen.
Cuando se lanzó World Community Grid, utilizaron el cliente propietario Grid MP de United Devices . Después de agregar soporte para el cliente BOINC de código abierto en 2005, World Community Grid eventualmente descontinuó el cliente Grid MP y se consolidó en la plataforma BOINC en 2008.
Aunque WCG utiliza software cliente de código abierto, es posible que las aplicaciones reales que realizan los cálculos científicos no lo sean. Sin embargo, varias de las aplicaciones científicas están disponibles con una licencia gratuita, aunque la fuente no está disponible directamente en WCG.
Problemas potenciales
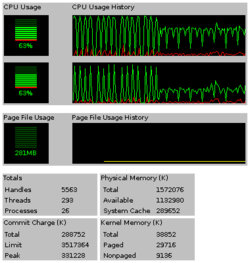
El software World Community Grid aumenta el uso de la CPU al consumir tiempo de procesamiento no utilizado; a finales de la década de 1990 y principios de la de 2000, estos cálculos estaban destinados a reducir los ciclos de CPU "desperdiciados". Con las CPU modernas, donde prevalece el escalado de frecuencia dinámico , el mayor uso hace que el procesador funcione a una frecuencia más alta, lo que aumenta el uso de energía y el calentamiento contra la administración de energía . Además, debido a un enfoque cada vez mayor en el rendimiento de la energía, o el rendimiento por vatio , la conexión de computadoras viejas / ineficientes a la red aumentará la energía total / promedio requerida para completar los mismos cálculos.
El cliente BOINC evita ralentizar la computadora mediante el uso de una variedad de límites que suspenden el cálculo cuando no hay suficientes recursos libres. A diferencia de otros proyectos BOINC, World Community Grid estableció los valores predeterminados de BOINC de manera conservadora, lo que hace que las posibilidades de daños en la computadora sean extremadamente pequeñas. El acelerador predeterminado de la CPU es del 60%. El acelerador es de grano grueso; por ejemplo, si el uso se establece en 60%, funcionará al 100% durante 3 segundos, luego al 0% durante 2 segundos, lo que dará como resultado una disminución promedio del uso del procesador.
Un programa complementario para computadoras con Windows, TThrottle , puede resolver el problema del sobrecalentamiento limitando directamente el uso de la computadora host por parte del proyecto BOINC. Lo hace midiendo la temperatura de la CPU y / o GPU y ajusta el tiempo de ejecución en consecuencia. También utiliza un tiempo de conmutación más corto de menos de un segundo, lo que resulta en un menor cambio de temperatura durante la conmutación.
Estadísticas y competencia
Las contribuciones de cada usuario se registran y las estadísticas de contribución de los usuarios están disponibles públicamente. Debido al hecho de que el tiempo de procesamiento de cada unidad de trabajo varía de una computadora a otra, dependiendo de la dificultad de la unidad de trabajo, la velocidad de la computadora y la cantidad de recursos inactivos disponibles, las contribuciones generalmente se miden en términos de puntos . Se otorgan puntos por cada unidad de trabajo en función del esfuerzo requerido para procesarla.
Al completar una unidad de trabajo, el cliente de BOINC solicitará la cantidad de puntos que cree que merece en función de los puntos de referencia del software ( consulte BOINC Credit System # Cobblestones ). Dado que varias computadoras procesan la misma unidad de trabajo para garantizar la precisión, los servidores de World Community Grid pueden ver los puntos reclamados por cada una de esas computadoras. Los servidores de WCG ignoran los valores atípicos estadísticos, promedian los valores restantes y otorgan el número resultante de puntos a cada computadora.
Dentro de la cuadrícula, los usuarios pueden unirse a equipos que han sido creados por organizaciones, grupos o individuos. Los equipos permiten un mayor sentido de identidad comunitaria y también pueden inspirar competencia. A medida que los equipos compiten entre sí, se realiza más trabajo para la parrilla en general.
Superar a
World Community Grid reconoce a las empresas y organizaciones como socios si promueven WCG dentro de su empresa u organización. En abril de 2021, WCG tenía 452 socios.
Además, como parte de su compromiso de mejorar la salud y el bienestar humanos, los resultados de todos los cálculos completados en World Community Grid se publican en el dominio público y se ponen a disposición de la comunidad científica.
Resultados científicos
Desde su lanzamiento, más de treinta proyectos se han ejecutado en World Community Grid. Algunos de los resultados incluyen:
- En febrero de 2014, los científicos del proyecto Help Fight Childhood Cancer anunciaron el descubrimiento de 7 compuestos que destruyen las células cancerosas del neuroblastoma sin efectos secundarios aparentes. Este descubrimiento, realizado con el apoyo de los voluntarios de WCG, es un paso positivo hacia un nuevo tratamiento. El proyecto ha anunciado que busca una colaboración con una empresa farmacéutica para desarrollar los compuestos en tratamientos. Dado el éxito del proyecto, los científicos han declarado que ya están planificando un proyecto de seguimiento que se centrará en otros cánceres pediátricos , posiblemente en colaboración con un grupo de oncología panasiático recién formado, del que son miembro fundador.
- En julio de 2012, el Proyecto de plegado del proteoma humano ha publicado varios artículos utilizando datos de WCG. Estos incluyen un documento sobre métodos de validación y una nueva base de datos de predicciones de estructura y función de proteínas; un artículo sobre la identificación de proteínas que regulan los procesos humanos; un artículo sobre el análisis de los genomas de cinco familias de plantas y sus proteomas, para el que se utilizó WCG en la creación de más de 29.000 estructuras de proteínas; un artículo sobre el proteoma de Saccharomyces cerevisiae .
- El proyecto GO Fight Against Malaria informó del descubrimiento de varias moléculas que son eficaces contra la malaria y la tuberculosis resistente a los medicamentos (incluida la TDR-TB, para la que no hay tratamiento disponible). El proyecto también probó nuevas moléculas contra MRSA , filariasis y peste bubónica . Continúan las pruebas de laboratorio para convertir esas moléculas en posibles tratamientos. GFAM también fue el primer proyecto en realizar mil millones de cálculos de acoplamiento diferentes. Se publicó un artículo en enero de 2015, con dos más pendientes de presentación. En junio de 2015, el proyecto informó que de los dos "éxitos" descubiertos contra una cepa de tuberculosis resistente a los medicamentos, se han sintetizado varios "análogos", el mejor de los cuales inhibe el crecimiento de Mycobacterium tuberculosis y es relativamente no tóxico para los mamíferos. células. La falta de financiación impidió seguir investigando los datos.
- Los científicos del proyecto Discovering Dengue Drugs - Together informaron del descubrimiento de varios nuevos inhibidores de la proteasa del dengue, la mayoría de los cuales también inhiben la proteasa del virus del Nilo Occidental . Un puñado de estos ya ha entrado en "estudios de eficacia y farmacocinética preclínicos cruciales". En noviembre de 2014, una actualización informó que los científicos tienen un fármaco que desactiva una enzima clave que permite que el virus del dengue se replique. También ha mostrado el mismo comportamiento en otros flavivirus, como el virus del Nilo Occidental. No se han observado efectos secundarios negativos como toxicidad, carcinogenicidad o mutagenicidad, lo que convierte a este fármaco en un candidato a fármaco antivírico muy potente para estos virus. Los científicos ahora están trabajando para sintetizar variantes de la molécula para mejorar su actividad y entrar en ensayos clínicos y preclínicos planificados. Sin embargo, en una actualización de octubre de 2018, el equipo de investigación informó que ninguno de sus diseños actuales había producido un inhibidor de la proteasa del dengue altamente potente que pudiera probarse in vivo .
- En junio de 2013, el Clean Energy Project publicó una base de datos de más de 2,3 millones de moléculas orgánicas cuyas propiedades se han caracterizado. De estas, 35.000 moléculas han mostrado el potencial de duplicar la eficiencia con respecto a las células solares orgánicas que se están produciendo actualmente. Antes de esta iniciativa, los científicos conocían solo un puñado de materiales a base de carbono que podían convertir la luz solar en electricidad de manera eficiente.
- En febrero de 2010, los científicos del proyecto FightAIDS @ Home anunciaron que habían encontrado dos compuestos que hacen posible una clase potencialmente nueva de medicamentos para combatir el SIDA. Los compuestos se adhieren al virus en sitios de unión recién descubiertos y, por lo tanto, pueden usarse para "mejorar las terapias existentes, tratar cepas de la enfermedad resistentes a los medicamentos y ralentizar la evolución de la resistencia a los medicamentos en el virus".
- En julio de 2015, el proyecto de búsqueda de medicamentos para la leishmaniasis anunció que había probado los 10 compuestos principales con la mayor eficiencia prevista de más de 100 identificados a través de unidades de trabajo WCG. De esos 10, 4 mostraron "resultados positivos" en las pruebas in vitro , y uno mostró "un resultado excepcionalmente prometedor". En agosto de 2017, las pruebas in vivo de los 4 compuestos en hámsteres mostraron resultados favorables, con un compuesto que indujo "una curación casi completa de las lesiones en dos de cada cinco hámsteres". Sin embargo, en una actualización de marzo de 2018, el equipo de investigación anunció que ninguno de los 10 compuestos probados tenía suficiente actividad anti-leishmaniasis.
- En julio de 2015, el proyecto Computing for Clean Water anunció que se había publicado un artículo en la revista Nature Nanotechnology que describe un nuevo tipo de filtro de agua que utiliza nanotubos de manera eficiente. "[Los] nanotubos están hechos de láminas de átomos de carbono de un solo átomo de espesor, llamadas grafeno , enrolladas en pequeños tubos, con diámetros de unos pocos nanómetros, una diezmilésima parte del diámetro de un cabello humano. El tamaño del Los tubos permiten el paso de las moléculas de agua, pero bloquean los patógenos y contaminantes más grandes, purificando el agua ". Al ejecutar simulaciones en WCG, los científicos descubrieron que ciertos tipos de vibraciones naturales llamadas fonones , en condiciones específicas, pueden conducir a un aumento de más del 300% en el flujo de agua a través de los nanotubos, en comparación con las predicciones teóricas anteriores.
- En abril de 2015, los científicos del proyecto Say No To Schistosoma informaron que se habían realizado análisis posteriores y se habían identificado las tres sustancias candidatas más prometedoras para las pruebas in vitro .
- En marzo de 2019, los investigadores de FightAIDS @ Home publicaron un artículo que describe una "Nueva interacción entre subunidades crítica para el ensamblaje principal del VIH-1" que "define un bolsillo de unión de inhibidores potencialmente objetivo". Utilizando World Community Grid, se utilizaron más de 1,6 millones de compuestos para apuntar a 20 conformaciones de este bolsillo. Los resultados preliminares sugieren que es un sitio de unión plausible para compuestos antivirales. El análisis adicional de estos compuestos es objeto de un estudio independiente.
Proyectos activos
OpenPandemics - COVID-19
El 1 de abril de 2020, IBM anunció OpenPandemics - COVID-19 . El proyecto tiene como objetivo identificar posibles tratamientos para el síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) que es responsable de la pandemia COVID-19 . WCG se asociará con Scripps Research , con quien se ha asociado en el pasado, especialmente en los proyectos FightAIDS @ Home . El proyecto se ejecuta en CPU y GPU y también servirá para crear una "herramienta de código abierto de respuesta rápida que ayudará a todos los científicos a buscar rápidamente tratamientos para pandemias futuras".
El proyecto se lanzó el 14 de mayo de 2020.
Mapeo de marcadores de cáncer
Mapeo de marcadores de cáncer (lanzado el 8 de noviembre de 2013). El proyecto tiene como objetivo identificar los marcadores asociados con varios tipos de cáncer y está analizando millones de puntos de datos recopilados de miles de muestras de tejido de pacientes sanos y cancerosos. Estos incluyen tejidos con cánceres de pulmón, ovario, próstata, páncreas y mama. Al comparar estos diferentes puntos de datos, los investigadores tienen como objetivo identificar patrones de marcadores para diferentes cánceres y correlacionarlos con diferentes resultados, incluida la capacidad de respuesta a varias opciones de tratamiento. El proyecto se centra en 4 tipos de cáncer, el primero de los cuales se centra en el cáncer de pulmón, y pasará al cáncer de ovario, el cáncer de próstata y el sarcoma.
FightAIDS @ Home Fase 2
FightAIDS @ Home Phase 2 (lanzado el 30 de septiembre de 2015) analiza más de cerca los resultados de la Fase 1. El proyecto tiene dos objetivos en los primeros experimentos; la arquitectura de simulación está funcionando correctamente y brinda resultados confiables, y el uso de BEDAM y AutoDock juntos brinda mejores resultados que usar solo BEDAM o AutoDock.
Ayude a detener la tuberculosis
Help Stop TB se lanzó en marzo de 2016 para ayudar a combatir la tuberculosis , una enfermedad causada por una bacteria que está desarrollando resistencia a los tratamientos actualmente disponibles. Los cálculos de este proyecto apuntan a los ácidos micólicos en la capa protectora de la bacteria, simulando el comportamiento de estas moléculas para comprender mejor cómo ofrecen protección a las bacterias.
Aplastar el cáncer infantil
Lanzado en enero de 2017, el proyecto Smash Childhood Cancer se basa en el trabajo del proyecto Help Fight Childhood Cancer mediante la búsqueda de candidatos a fármacos dirigidos a cánceres infantiles adicionales. Tras el retiro del Dr. Akira Nakagawara en marzo de 2020, el investigador principal cambió al Dr. Godfrey Chan, quien era uno de los miembros originales del equipo de Smash Childhood Cancer. Además, se han agregado PRDM14 y Fox01 como nuevos objetivos de investigación.
Proyecto de lluvia en África
El Africa Rainfall Project (lanzado en octubre de 2019) utilizará la potencia informática de World Community Grid, los datos de The Weather Company y otros datos para mejorar el modelado de las precipitaciones, lo que puede ayudar a los agricultores del África subsahariana a cultivar con éxito sus cultivos.
Proyectos completados
Plegado del proteoma humano - Fase 1
El primer proyecto lanzado en World Community Grid fue Human Proteome Folding Project, o HPF1, que tiene como objetivo predecir la estructura de las proteínas humanas . El proyecto se lanzó el 16 de noviembre de 2004 y se completó el 18 de julio de 2006. Este proyecto fue único en el sentido de que el cálculo se realizó en conjunto con el proyecto de computación distribuida grid.org . Diseñado por Richard Bonneau en el Instituto de Biología de Sistemas , el proyecto utilizó la computación en cuadrícula para producir las estructuras probables para cada una de las proteínas utilizando una puntuación de Rosetta. A partir de estas predicciones, los investigadores esperan predecir la función de la miríada de proteínas. Esta mayor comprensión de las proteínas humanas podría resultar vital en la búsqueda de curas para enfermedades humanas . La computación para este proyecto se completó oficialmente el 18 de julio de 2006. Se han publicado los resultados de la investigación para la porción de levadura de HPF1.
Plegado del proteoma humano - Fase 2
Plegado del proteoma humano - Fase 2 (HPF2) (lanzado el 23 de junio de 2006) fue el tercer proyecto que se ejecutó en World Community Grid y se completó en 2013. Este proyecto, que sigue a HPF1, se centró en las proteínas secretadas por humanos , con especial atención en biomarcadores y proteínas en la superficie de las células, así como en Plasmodium , el organismo que causa la malaria. HPF2 genera modelos de proteínas de mayor resolución que HPF1. Aunque estos modelos de mayor resolución son más útiles, también requieren más potencia de procesamiento para generar.
En un informe de estado de julio de 2012, los científicos del proyecto informaron que el Dr. Markus Landthaler, del Centro Max Delbruch de Medicina Molecular (MDC) de Berlín, estaba utilizando los resultados generados por los cálculos de WCG. Los resultados de HPF2 ayudaron al Dr. Markus Landthaler y sus colaboradores a redactar un nuevo artículo sobre "El proteoma ligado al ARNm y su perfil de ocupación global en las transcripciones de codificación de proteínas"
Ayuda a derrotar al cáncer
El proyecto Help Defeat Cancer busca mejorar la capacidad de los profesionales médicos para determinar las mejores opciones de tratamiento para pacientes con cáncer de mama, cabeza o cuello. El proyecto se lanzó el 20 de julio de 2006 y se completó en abril de 2007. El proyecto funcionó identificando patrones visuales en un gran número de microarrays de tejido tomados de muestras de tejido archivadas. Al correlacionar los datos del patrón con la información sobre el tratamiento y el resultado del paciente, los resultados de este proyecto podrían ayudar a proporcionar mejores opciones de tratamiento específicas.
Comparación del genoma
El proyecto Genome Comparison está patrocinado por la institución brasileña de investigación Fiocruz . El proyecto se lanzó el 21 de noviembre de 2006 y se completó el 21 de julio de 2007. El proyecto busca comparar secuencias de genes de diferentes organismos entre sí para encontrar similitudes entre ellos. Los científicos esperan descubrir para qué sirve una secuencia de genes en particular en una función particular de un organismo, comparándola con una secuencia de genes similar de función conocida en otro organismo.
Ayude a curar la distrofia muscular - Fase 1
Help Cure Muscular Dystrophy está dirigido por Décrypthon , una colaboración entre la Asociación Francesa de Distrofia Muscular, el Centro Nacional Francés de Investigación Científica e IBM . La Fase 1 se lanzó el 19 de diciembre de 2006 y se completó el 11 de junio de 2007. El proyecto investigó las interacciones proteína-proteína de 40.000 proteínas cuyas estructuras son conocidas, con especial atención a aquellas proteínas que desempeñan un papel en las enfermedades neuromusculares . La base de datos de la información producida ayudará a los investigadores a diseñar moléculas para inhibir o mejorar la unión de macromoléculas particulares , lo que con suerte conducirá a mejores tratamientos para la distrofia muscular y otras enfermedades neuromusculares. Este proyecto solo estaba disponible para los agentes que ejecutaban el cliente Grid MP , por lo que no estaba disponible para los usuarios que ejecutaban BOINC .
Descubriendo los medicamentos contra el dengue, juntos
Discovering Dengue Drugs - Together fue patrocinado por científicos de la Universidad de Texas y la Universidad de Chicago y se desarrollará en dos fases. La fase 1, lanzada el 21 de agosto de 2007, utilizó AutoDock 2007 (el mismo software que se utiliza para FightAIDS @ Home ) para probar posibles fármacos antivirales (mediante la inhibición de la proteasa NS3 ) contra virus de la familia flaviviridae y se completó el 11 de agosto de 2009. Fase 2 " [utiliza] un programa de computación más intensivo para seleccionar a los candidatos que pasan por la Fase 1. " Los candidatos a fármacos que pasen por la Fase 2 se someterán a pruebas de laboratorio.
AfricanClimate @ Home
La misión de AfricanClimate @ Home era desarrollar modelos climáticos más precisos de regiones específicas de África. Se pretendía que sirviera de base para comprender cómo cambiará el clima en el futuro, de modo que se pudieran implementar medidas diseñadas para aliviar los efectos adversos del cambio climático. La tremenda potencia informática de World Community Grid se utilizó para comprender y reducir la incertidumbre con la que se simulaban los procesos climáticos en África. La fase 1 de African Climate @ Home se lanzó el 3 de septiembre de 2007 y finalizó en julio de 2008.
Ayuda a conquistar el cáncer
El proyecto Help Conquer Cancer (lanzado el 1 de noviembre de 2007) está patrocinado por el Ontario Cancer Institute (OCI), el Princess Margaret Hospital y la University Health Network de Toronto, Canadá. El proyecto consiste en cristalografía de rayos X . La misión de Help Conquer Cancer es mejorar los resultados de la cristalografía de rayos X de proteínas, que ayuda a los investigadores no solo a anotar partes desconocidas del proteoma humano, sino que mejora de manera importante su comprensión del inicio, la progresión y el tratamiento del cáncer.
El proyecto HCC fue el primer proyecto WCG que se benefició de las unidades de procesamiento de gráficos (GPU) que ayudaron a terminarlo mucho antes de lo proyectado inicialmente debido a la enorme potencia de las GPU. En el informe de estado de abril de 2013, los científicos informan que todavía hay muchos datos por analizar, pero que están preparando un nuevo proyecto que buscará firmas pronósticas y predictivas (conjuntos de genes, proteínas, microARN, etc.) que ayuden a predecir al paciente. supervivencia y respuesta al tratamiento. El proyecto finalizó en mayo de 2013.
Arroz nutritivo para el mundo
El Arroz Nutritivo para el Mundo proyecto se lleva a cabo por Ram Samudrala 's Biología Computacional Grupo de Investigación en la Universidad de Washington . El proyecto se lanzó el 12 de mayo de 2008 y se completó el 6 de abril de 2010. El propósito de este proyecto es predecir la estructura de las proteínas de las principales cepas de arroz , con el fin de ayudar a los agricultores a producir mejores cepas de arroz con mayores rendimientos de cultivos . promover una mayor resistencia a las enfermedades y plagas , y utilizar una gama completa de nutrientes biodisponibles que pueden beneficiar a las personas en todo el mundo, especialmente en regiones donde la desnutrición es una preocupación crítica. El proyecto ha sido cubierto por más de 200 medios de comunicación desde su inicio. El 13 de abril de 2010, World Community Grid anunció oficialmente que el proyecto Arroz nutritivo para el mundo finalizó el 6 de abril de 2010.
En abril de 2014, se publicó una actualización que indicaba que el equipo de investigación pudo publicar información estructural sobre miles de proteínas y avanzar en el campo del modelado computacional de proteínas. Se espera que estos resultados, que solo fueron posibles debido a la enorme cantidad de potencia informática donada que tenían disponible, guíen los esfuerzos futuros de investigación y ciencia de las plantas.
El proyecto de energía limpia
El proyecto de Energía Limpia está patrocinado por científicos del Departamento de Química y Biología Química de la Universidad de Harvard . La misión del Clean Energy Project es encontrar nuevos materiales para la próxima generación de células solares y, posteriormente, dispositivos de almacenamiento de energía . Los investigadores están empleando la mecánica molecular y los cálculos de estructuras electrónicas para predecir las propiedades ópticas y de transporte de las moléculas que podrían convertirse en la próxima generación de materiales de células solares.
La Fase 1 se lanzó el 5 de diciembre de 2008 y se completó el 13 de octubre de 2009. Al aprovechar la potencia de cálculo de World Community Grid, los investigadores pudieron calcular las propiedades electrónicas de decenas de miles de materiales orgánicos, muchos más que nunca. ser probado en un laboratorio y determinar qué candidatos son más prometedores para desarrollar tecnología de energía solar asequible .
La Fase 2 se lanzó el 28 de junio de 2010, patrocinada por los científicos del Departamento de Química y Biología Química de la Universidad de Harvard . Se están realizando más cálculos sobre las propiedades ópticas, electrónicas y otras propiedades físicas de los materiales candidatos con el software de química cuántica Q-Chem . Sus hallazgos se han enviado a la revista Energy & Environmental Science .
Ayude a combatir el cáncer infantil
El proyecto Help Fight Childhood Cancer (lanzado el 13 de marzo de 2009) está patrocinado por científicos del Instituto de Investigación del Centro de Cáncer de Chiba y la Universidad de Chiba . La misión del proyecto Help Fight Childhood Cancer es encontrar medicamentos que puedan inhabilitar tres proteínas particulares asociadas con el neuroblastoma , uno de los tumores sólidos más frecuentes en los niños. La identificación de estos medicamentos podría potencialmente hacer que la enfermedad sea mucho más curable cuando se combina con un tratamiento de quimioterapia .
Búsqueda de medicamentos antivirales contra la influenza
El proyecto de búsqueda de medicamentos antivirales contra la influenza está patrocinado por el Dr. Stan Watowich y su equipo de investigación en la rama médica de la Universidad de Texas ( Galveston , Texas , EE. UU.). El proyecto se lanzó el 5 de mayo de 2009 y se completó el 22 de octubre de 2009. La misión del proyecto Búsqueda de medicamentos antivirales contra la influenza es encontrar nuevos medicamentos que puedan detener la propagación de una infección por influenza en el cuerpo. La investigación abordará específicamente las cepas de influenza que se han vuelto resistentes a los medicamentos, así como las nuevas cepas que están apareciendo. La identificación de los compuestos químicos que son los mejores candidatos acelerará los esfuerzos para desarrollar tratamientos que serían útiles en el manejo de brotes de influenza estacional y futuras epidemias e incluso pandemias de influenza. La fase 1 del proyecto de búsqueda de medicamentos antivirales contra la influenza ya finalizó el 22 de octubre de 2009. Ahora los investigadores están realizando el posprocesamiento de los resultados de la fase 1 y se están preparando para la fase 2.
En noviembre de 2012, los científicos del proyecto declararon que, dado que no existe un peligro inmediato de un brote de influenza, todos los resultados del proyecto se publicarían en línea y sus recursos se reenfocarían en el Proyecto Dengue.
Ayude a curar la distrofia muscular - Fase 2
World Community Grid e investigadores apoyados por Decrypthon, una asociación entre AFM (Asociación Francesa de Distrofia Muscular), CNRS (Centro Nacional Francés de Investigación Científica), Universite Pierre et Marie Curie e IBM estaban investigando interacciones proteína-proteína para más de 2200 proteínas cuyos Se conocen estructuras, con especial atención a aquellas proteínas que desempeñan un papel en las enfermedades neuromusculares . La Fase 2 se lanzó el 12 de mayo de 2009 y se completó el 26 de septiembre de 2012. La base de datos de información producida ayudará a los investigadores a diseñar moléculas para inhibir o mejorar la unión de macromoléculas particulares , lo que con suerte conducirá a mejores tratamientos para la distrofia muscular y otras enfermedades neuromusculares.
La fase 2 del proyecto Help Cure Muscular Dystrophy comenzó una vez analizados los resultados de la primera fase. La fase 2 se ejecutó en la plataforma BOINC .
Descubriendo los medicamentos contra el dengue - Juntos - Fase 2
Discovering Dengue Drugs - Together - Phase 2 (lanzado el 17 de febrero de 2010) está patrocinado por la rama médica de la Universidad de Texas (UTMB) en Galveston , Texas , Estados Unidos y la Universidad de Chicago en Illinois , Estados Unidos. La misión es identificar candidatos a fármacos prometedores para combatir el dengue , la hepatitis C , el Nilo Occidental , la fiebre amarilla y otros virus relacionados. La amplia potencia informática de World Community Grid se utilizará para completar los cálculos de descubrimiento de fármacos basados en la estructura necesarios para identificar estos candidatos a fármacos.
Computación para agua limpia
Computing for Clean Water (lanzado el 20 de septiembre de 2010) está patrocinado por el Centro de Nano y Micro Mecánica de la Universidad de Tsinghua en Beijing . La misión del proyecto es proporcionar una visión más profunda a escala molecular de los orígenes del flujo eficiente del agua a través de una nueva clase de materiales filtrantes. Esta información, a su vez, guiará el desarrollo futuro de filtros de agua más eficientes y de bajo costo. Se estima que 1.200 millones de personas carecen de acceso a agua potable y 2.600 millones tienen poco o ningún saneamiento. Como resultado, millones de personas mueren anualmente, aproximadamente 3.900 niños al día debido a la falta de agua potable. El 25 de abril de 2014, los científicos del proyecto publicaron una actualización en la que indicaban que tenían resultados interesantes que informar cuando se presentaba el documento y que el proyecto sobre WCG estaba terminado.
Búsqueda de medicamentos para la leishmaniasis
La búsqueda de medicamentos para la leishmaniasis (lanzada el 7 de septiembre de 2011) está encabezada por la Universidad de Antioquia en Medellín , Colombia , con la asistencia de investigadores de la Rama Médica de la Universidad de Texas en Galveston, Texas. La misión es identificar posibles candidatos a moléculas que posiblemente podrían desarrollarse en tratamientos para la leishmaniasis . El extenso poder de computación de World Community Grid se utilizará para realizar simulaciones por computadora de las interacciones entre millones de compuestos químicos y ciertas proteínas objetivo. Esto ayudará a encontrar los compuestos más prometedores que pueden conducir a tratamientos efectivos para la enfermedad.
Proyecto GO Fight Against Malaria
La misión del proyecto GO Fight Against Malaria (lanzado el 16 de noviembre de 2011) es descubrir candidatos a fármacos prometedores que puedan convertirse en nuevos fármacos que curen las formas de malaria resistentes a los fármacos . El poder de computación de World Community Grid se utilizará para realizar simulaciones por computadora de las interacciones entre millones de compuestos químicos y ciertas proteínas objetivo, para predecir su capacidad para eliminar la malaria. Los mejores compuestos serán probados por científicos del Instituto de Investigación Scripps de La Jolla, California, EE. UU. Y se desarrollarán aún más en posibles tratamientos para la enfermedad.
Di no al esquistosoma
Say No to Schistosoma (lanzado el 22 de febrero de 2012) fue el vigésimo proyecto de investigación que se lanzó en World Community Grid. Los investigadores de la Universidad Informaium en Belo Horizonte y FIOCRUZ-Minas , Brasil , ejecutaron este proyecto en World Community Grid para realizar simulaciones por computadora de las interacciones entre millones de compuestos químicos y ciertas proteínas diana con la esperanza de encontrar tratamientos efectivos para la esquistosomiasis . En abril de 2015, se habían realizado análisis posteriores y se habían identificado las tres sustancias candidatas más prometedoras para las pruebas in vitro.
Computación para agua sostenible
Computing for Sustainable Water fue el vigésimo primer proyecto de investigación que se lanzó en World Community Grid. Los investigadores de la Universidad de Virginia estaban ejecutando este proyecto en World Community Grid para estudiar los efectos de la actividad humana en una gran cuenca hidrográfica y obtener conocimientos más profundos sobre qué acciones pueden respaldar la restauración, la salud y la sostenibilidad de este importante recurso hídrico. El proyecto se lanzó el 17 de abril de 2012 y se completó el 17 de octubre de 2012.
Descubriendo los misterios del genoma
El proyecto Uncovering Genome Mysteries se lanzó el 16 de octubre de 2014 y es una colaboración conjunta entre científicos australianos y brasileños. El proyecto tiene como objetivo examinar cerca de 200 millones de genes de muchas formas de vida y compararlos con genes conocidos para averiguar cuál es su función. Los resultados podrían tener un efecto en campos como la medicina y la investigación medioambiental.
Juntos ser más astutos que el ébola
Outsmart Ebola Together fue una colaboración con el Instituto de Investigación Scripps para ayudar a encontrar compuestos químicos para combatir la enfermedad por el virus del Ébola . Fue lanzado el 3 de diciembre de 2014. El objetivo es bloquear pasos cruciales en el ciclo de vida del virus, mediante la búsqueda de fármacos con alta afinidad de unión con algunas de sus proteínas. Hay dos objetivos: una proteína de superficie utilizada por el virus para infectar células humanas y proteínas "transformadoras" que cambian de forma para llevar a cabo diferentes funciones. El proyecto se completó oficialmente el 6 de diciembre de 2018.
OpenZika
OpenZika se lanzó el 18 de mayo de 2016 para ayudar a combatir el virus Zika . El proyecto se centra en las proteínas que se cree que utiliza el virus Zika para sobrevivir y diseminarse en el cuerpo, según los resultados conocidos de enfermedades similares como el dengue y la fiebre amarilla . Estos resultados ayudarán a los investigadores a desarrollar un medicamento contra el Zika. El proyecto se completó oficialmente el 13 de diciembre de 2019.
FightAIDS @ Home
FightAIDS @ Home (lanzado el 19 de noviembre de 2005) fue el segundo proyecto de World Community Grid y el primero en atacar una sola enfermedad. Cada computadora individual procesa una molécula de fármaco potencial y prueba qué tan bien encajaría con la proteasa del VIH , actuando como un inhibidor de la proteasa . El Instituto de Investigación Scripps publicó su primer artículo científico revisado por pares sobre los resultados de FightAIDS @ Home el 21 de abril de 2007. Este artículo explica que los resultados hasta ese momento se utilizarán principalmente para mejorar la eficiencia de los cálculos futuros de FightAIDS @ Home.
Proyecto de inmunidad del microbioma
Microbiome Immunity Project (lanzado en agosto de 2017) es un estudio de proteínas en bacterias ubicadas dentro y sobre el cuerpo humano; el microbioma humano , que está compuesto por alrededor de 3 millones de genes bacterianos separados. Al aprender los genes de las bacterias, se pueden conocer sus formas individuales y cada forma física determina la función de las bacterias. Las instituciones colaborativas incluyen la Universidad de California en San Diego , el Instituto Broad del MIT y Harvard, y el Instituto Flatiron de la Fundación Simons .
Ver también
- BOINC
- Plegable en casa
- Problema global
- Lista de proyectos de computación distribuida
- Informática voluntaria
- Comunidad mundial
Referencias
enlaces externos
- Cuadrícula de comunidad mundial
- Historia de Voice of America (junio de 2008)
- The New York Times> Tecnología> Potencia de PC no utilizada para ejecutar Grid para desenredar enfermedades
- Proyectos de voluntariado de mundos virtuales basados en la comunidad del universo virtual y la computación en red
