ARN ribosómico 5S - 5S ribosomal RNA
| ARN ribosómico 5S | |
|---|---|
| |
| Identificadores | |
| Símbolo | 5S_rRNA |
| Rfam | RF00001 CL00113 |
| Otros datos | |
| Tipo de ARN | Gene ; ARNr |
| Dominio (s) | Eucariota ; Bacterias ; Arqueas |
| VAMOS | IR: 0005840 IR: 0003735 |
| ASI QUE | Entonces: 0000652 |
| Estructuras PDB | PDBe |
El ARN ribosómico 5S ( ARNr 5S ) es una molécula de ARN ribosómico de aproximadamente 120 nucleótidos de longitud con una masa de 40 kDa . Es un componente estructural y funcional de la gran subunidad del ribosoma en todos los dominios de la vida ( bacterias , arqueas y eucariotas ), con la excepción de los ribosomas mitocondriales de hongos y animales . La designación 5S se refiere a la velocidad de sedimentación de la molécula en una ultracentrífuga, que se mide en unidades de Svedberg (S).

Biosíntesis
En los procariotas, el gen del ARNr 5S se localiza típicamente en los operones del ARNr aguas abajo del ARNr de las subunidades pequeña y grande , y se cotranscribe en un precursor policistrónico . Una particularidad de los genomas nucleares eucariotas es la aparición de múltiples copias del gen 5S rRNA (5S rDNA) agrupadas en repeticiones en tándem, con un número de copias que varía de una especie a otra. Eukaryotic 5S rRNA es sintetizada por la ARN polimerasa III , mientras que otros rRNAs eucariotas se escinden de un precursor 45S transcritos por la ARN polimerasa I . En los ovocitos de Xenopus , se ha demostrado que los dedos 4 a 7 del factor de transcripción TFIIIA de nueve dedos de zinc pueden unirse a la región central del ARN 5S. La unión entre el ARNr 5S y el TFIIIA sirve tanto para reprimir la transcripción adicional del gen del ARN 5S como para estabilizar la transcripción del ARN 5S hasta que se requiera para el ensamblaje del ribosoma.
Estructura
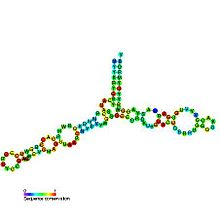
La estructura secundaria del ARNr 5S consta de cinco hélices (indicadas como IV en números romanos ), cuatro bucles (BE) y una bisagra (A), que forman juntas una estructura en forma de Y. Los bucles C y D son horquillas terminales y los bucles B y E son internos. Según los estudios filogenéticos, es probable que las hélices I y III sean ancestrales. La hélice III incluye dos adenosinas altamente conservadas. Se cree que Helix V, con su estructura de horquilla, interactúa con TFIIIA.
Ubicación dentro del ribosoma

Usando una variedad de técnicas moleculares, incluyendo la microscopía inmunoelectrónica , crio-microscopía electrónica , intermolecular química de reticulación , y cristalografía de rayos X , la ubicación de la 5S rRNA dentro de la subunidad ribosómica grande se ha determinado que una gran precisión. En bacterias y arqueas , la subunidad ribosómica grande (LSU) en sí misma está compuesta por dos restos de ARN, el ARNr 5S y otro ARN más grande conocido como ARNr 23S , junto con numerosas proteínas asociadas.
En eucariotas, la LSU contiene ARNr 5S, 5.8S y 28S e incluso más proteínas. La estructura de LSU en 3 dimensiones muestra una superficie relativamente lisa y la superficie opuesta tiene tres proyecciones, en particular la protuberancia L1, la protuberancia central (CP) y el tallo L7 / L12. La protuberancia L1 y el tallo L7 / L12 están dispuestos lateralmente rodeando a CP. El ARNr 5S se encuentra en el CP y participa en la formación y estructura de esta proyección. Los otros componentes principales de la protuberancia central incluyen el ARNr 23S (o alternativamente 28S en eucariotas) y varias proteínas, incluidas L5, L18, L25 y L27.
Funciones ribosomales
La función exacta del ARNr 5S aún no está clara. En Escherichia coli , las deleciones del gen de ARNr 5S reducen la tasa de síntesis de proteínas y tienen un efecto perjudicial más profundo sobre la aptitud celular que las deleciones de un número comparable de copias de otros genes de ARNr ( 16S y 23S ). Los estudios cristalográficos indican que las proteínas de unión al ARNr 5S y otras proteínas de la protuberancia central de la LSU desempeñan un papel en la unión de los ARNt. Además, la proximidad topográfica y física entre el ARNr 5S y el ARNr 23S, que forma el centro de asociación de peptidil transferasa y GTPasa, sugiere que el ARNr 5S actúa como mediador entre los dos centros funcionales del ribosoma formando, junto con la unión del ARNr 5S proteínas y otros componentes de la protuberancia central, puentes entre subunidades y sitios de unión de tRNA.
Funciones en el ensamblaje ribosómico
En eucariotas, el ribosoma citosólico se ensambla a partir de cuatro ARNr y más de 80 proteínas. Una vez transcrito, los extremos 3 'del ARNr 5S son procesados por las exonucleasas Rex1p, Rex2p y Rex3p. Las subunidades ribosómicas 60S y 40S se exportan desde el núcleo al citoplasma donde se unen para formar el ribosoma 80S maduro y capaz de traducir . El momento exacto en que el rRNA 5S se integra en el ribosoma sigue siendo controvertido, pero en general se acepta que el rRNA 5S se incorpora a la partícula 90S, que es un precursor de la partícula 60S, como parte de un pequeño complejo de RNP independiente del ribosoma formado por 5S rRNA y proteína ribosómica L5.
Interacciones con proteínas
A continuación se enumeran varias proteínas importantes que interactúan con el ARNr 5S.
La proteína
La interacción del ARNr 5S con la proteína La evita que el ARN se degrade por exonucleasas en la célula. La proteína se encuentra en el núcleo de todos los organismos eucariotas y se asocia con varios tipos de ARN transcritos por ARN pol III. La proteína interactúa con estos ARN (incluido el ARNr 5S) a través de su tracto de oligouridina 3 ', lo que ayuda a la estabilidad y al plegamiento del ARN.
Proteína L5
En las células eucariotas, la proteína ribosómica L5 se asocia y estabiliza el ARNr 5S formando una partícula de ribonucleoproteína prerribosómica (RNP) que se encuentra tanto en el citosol como en el núcleo. La deficiencia de L5 evita el transporte de ARNr 5S al núcleo y da como resultado una disminución del ensamblaje ribosómico.
Otras proteínas ribosómicas
En procariotas, el ARNr 5S se une a las proteínas ribosómicas L5, L18 y L25, mientras que en eucariotas solo se sabe que el ARNr 5S se une a la proteína ribosómica L5 . En T. brucei , el agente causante de la enfermedad del sueño , el ARNr 5S interactúa con dos proteínas de unión a ARN estrechamente relacionadas, P34 y P37, cuya pérdida da como resultado un nivel global más bajo de ARNr 5S.
Presencia en los ribosomas de orgánulos
| El genoma mitocondrial permutado codifica el ARNr 5S | |
|---|---|
| Identificadores | |
| Símbolo | mtPerm-5S |
| Rfam | RF02547 CL00113 |
| Otros datos | |
| Tipo de ARN | Gene ; ARNr |
| Dominio (s) | Eucariota ; |
| VAMOS | IR: 0005840 IR: 0003735 |
| ASI QUE | Entonces: 0000652 |
| Estructuras PDB | PDBe |
Los mecanismos de traducción de mitocondrias y plastidios (orgánulos de origen bacteriano endosimbiótico) y sus parientes bacterianos comparten muchas características, pero también muestran marcadas diferencias. Los genomas de orgánulos codifican ARNr de SSU y LSU sin excepción, sin embargo, la distribución de los genes de ARNr 5S ( rrn5 ) es muy desigual. Rrn5 se identifica fácilmente y es común en los genomas de la mayoría de los plastidios. En contraste, la rrn5 mitocondrial inicialmente parecía estar restringida a plantas y un pequeño número de protistas. Los rRNA 5S orgánulos adicionales y más divergentes solo se identificaron con modelos de covarianza especializados que incorporan información sobre el sesgo pronunciado de composición de la secuencia y la variación estructural. Este análisis identificó genes de ARNr 5S adicionales no solo en los genomas mitocondriales de la mayoría de los linajes protistas , sino también en los genomas de ciertos apicoplastos (plástidos no fotosintéticos de protozoos patógenos como Toxoplasma gondii y Eimeria tenella ).
Los ARNr mitocondriales de 5S de la mayoría de las stramenopiles comprenden la mayor diversidad de estructuras secundarias. Los ARNr 5S mitocondriales permutados en las algas pardas representan el caso menos convencional, donde la hélice de cierre I, que de lo contrario une los extremos 5 'y 3' de la molécula, se reemplaza por una horquilla (cerrada) que da como resultado una unión de tres vías abierta.
La evidencia actual indica que el ADN mitocondrial de solo unos pocos grupos, especialmente animales , hongos , alveolados y euglenozoos, carece del gen. La protuberancia central , ocupada por lo demás por ARNr 5S y sus proteínas asociadas (ver Figura 2 ), fue remodelada de varias formas. En los ribosomas mitocondriales fúngicos, el rRNA 5S es reemplazado por secuencias de expansión de rRNA LSU. En los cinetoplastidos (euglenozoos), la protuberancia central está formada por completo de proteínas ribosomales mitocondriales evolutivamente novedosas. Por último, los ribosomas mitocondriales animales han cooptado un ARNt mitocondrial específico (Val en vertebrados) para sustituir el ARNr 5S faltante.
Ver también
Referencias
enlaces externos
- Página de ARN ribosómico 5S en Rfam
- 5SData Archivado el 27 de abril de 2010 en la Wayback Machine.
- 5S + Ribosomal + RNA en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- Ubicación del gen humano 5S_rRNA en UCSC Genome Browser .
- Halococcus morrhuae (archaebacterium) ARNr 5S

