Ataque al promotor - Promoter bashing
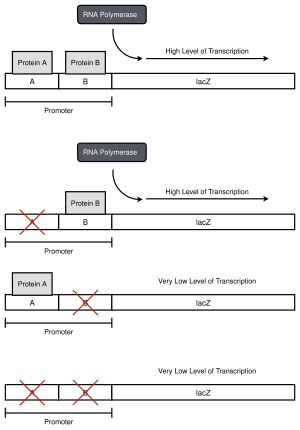
En un entorno de laboratorio, es posible que no se sepa que el promotor consta de dos regiones: se pueden realizar mutaciones únicas a lo largo del promotor, se puede secuenciar el promotor y analizar los niveles de informador para encontrar los límites para cada región.
El bashing de promotores es una técnica utilizada en biología molecular para identificar cómo ciertas regiones de una cadena de ADN , comúnmente promotores , afectan la transcripción de genes posteriores. En circunstancias normales, las proteínas se unen al promotor y activan o reprimen la transcripción. En un ensayo de bashing de promotores , se realizan mutaciones o deleciones puntuales específicas en regiones específicas del promotor y luego se mide la transcripción del gen. La contribución de una región del promotor se puede observar por el nivel de transcripción. Si una mutación o deleción cambia el nivel de transcripción, entonces se sabe que esa región del promotor puede ser un sitio de unión u otro elemento regulador.
El ataque al promotor se realiza a menudo con deleciones del extremo 5 ' o 3' de la cadena de ADN; este ensayo es más fácil de realizar basándose en la digestión de restricción repetida y en fragmentos de purificación en gel de tamaños específicos. A menudo es más fácil ligar el promotor en el indicador, generar una gran cantidad de la construcción del indicador mediante PCR o crecimiento en bacterias, y luego realizar digestiones de restricción en serie en esta muestra. La capacidad de los promotores cadena arriba puede ensayarse fácilmente eliminando segmentos del extremo 5 ', y lo mismo para el extremo 3' de la cadena para los promotores cadena abajo.
Como el promotor normalmente contiene secuencias de unión para proteínas que afectan la transcripción, esas proteínas también son necesarias cuando se prueban los efectos del promotor. Las proteínas que se asocian con el promotor pueden identificarse usando un ensayo de cambio de movilidad electroforética (EMSA), y los efectos de la inclusión o exclusión de las proteínas con los promotores mutagenizados pueden evaluarse en el ensayo. Esto permite que el uso del promotor bashing no solo descubra la ubicación en la hebra de ADN que afecta la transcripción, sino también las proteínas que afectan esa hebra. Los efectos de las interacciones de las proteínas entre sí, así como los sitios de unión, también pueden ensayarse de esta forma; En cambio, las proteínas candidatas deben identificarse mediante ensayos de interacción proteína / proteína en lugar de una EMSA.
Procedimiento
Este es un procedimiento de ejemplo para un ensayo de bashing de promotores, adaptado de Boulin et al. :
- Clone la región de ADN que se cree que actúa como promotora. La clonación es necesaria para el ensayo porque asegura que el promotor es el único factor que afecta la expresión. Este paso a menudo implica la extracción del ADN del organismo en el que reside y la amplificación por PCR .
- Secuencia de la región. La secuenciación de ADN es necesaria para identificar diferencias en los promotores mutados del promotor de tipo salvaje y para correlacionar esas diferencias con las diferencias en la expresión génica. Además, ayuda con la restricción de digestión de la región.
- Digerir con endonucleasas de restricción apropiadas. La región se puede digerir para eliminar elementos que se cree que no forman parte del promotor. Además, el gen indicador debe insertarse a una distancia determinada del promotor para la mayoría de los promotores. En algunos métodos de bashing de promotores, se utilizan digestiones de restricción múltiples para eliminar sistemáticamente elementos de los promotores; este método asegura que las regiones del promotor eliminadas no contribuyan a la expresión del informador .
- Mutagenizar el promotor. Es necesario mutar el promotor si no se usa el método de eliminar parte del promotor con digestión de restricción. Se pueden generar muchas hebras mutadas, secuenciar las hebras y ensayar las actividades de los promotores. Esto a menudo es necesario porque no se puede garantizar que una mutación inactive un sitio de unión. También se puede usar mutagénesis basada en PCR no dirigida; los parámetros de la reacción de PCR mutagénica se pueden ajustar para introducir un número razonable de mutaciones. Sin embargo, la naturaleza aleatoria de la PCR requiere que se analicen más cadenas después de este paso.
- Ligar al gen reportero. Los promotores que se van a ensayar deben ligarse a un gen indicador de modo que se puedan medir los niveles de expresión génica. El gen informador debe estar a una distancia suficiente del promotor para que el promotor lo afecte como un promotor de tipo salvaje afectaría a un gen. Esto se puede verificar con el control positivo (promotor completo).
- Transforma las células de interés con las diversas construcciones de promotor: informador. Las construcciones de promotor e informador deben ligarse en un plásmido y transformarse en células en las que ese plásmido puede expresarse para medir la actividad de cada secuencia promotora. Las proteínas que afectan al promotor también deben agregarse a esas células; a menudo, esas proteínas se colocan en el mismo plásmido o en uno diferente bajo la regulación de un promotor constitutivamente activo.
- Mide las tasas de transcripción del gen reportero. Se analizan los productos génicos y se miden las tasas de transcripción del informador.
A partir de los datos recibidos de analizar los diferentes promotores, se pueden determinar los efectos de varias partes del promotor. Sin embargo, es posible que no haya suficientes datos presentes y el ensayo deba volver a ejecutarse con una región promotora diferente y / o mutaciones diferentes.
