Conjunto de ciclo de polimerasa - Polymerase cycling assembly
El ensamblaje de ciclos de polimerasa (o PCA , también conocido como ensamblaje de PCR ) es un método para el ensamblaje de oligonucleótidos de ADN grandes a partir de fragmentos más cortos. El proceso utiliza la misma tecnología que la PCR , pero aprovecha la hibridación y el apareamiento del ADN, así como la ADN polimerasa para amplificar una secuencia completa de ADN en un orden preciso basado en los oligonucleótidos monocatenarios utilizados en el proceso. Por tanto, permite la producción de genes sintéticos e incluso genomas sintéticos completos .
Principios de la PCA
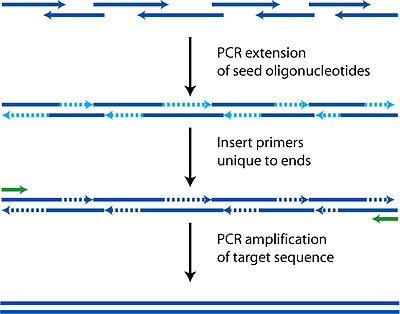
De manera muy similar a cómo se diseñan los cebadores de manera que haya un cebador directo y un cebador inverso capaces de permitir que la ADN polimerasa llene toda la secuencia de la plantilla, PCA usa la misma tecnología pero con múltiples oligonucleótidos. Mientras que en la PCR el tamaño habitual de los oligonucleótidos utilizados es de 18 pares de bases, en PCA se utilizan longitudes de hasta 50 para garantizar la unicidad y la hibridación correcta.
Cada oligonucleótido está diseñado para ser parte de la cadena superior o inferior de la secuencia diana. Además del requisito básico de tener que poder enlosar la secuencia diana completa, estos oligonucleótidos también deben tener las propiedades habituales de temperaturas de fusión similares, libres de horquillas y no demasiado ricos en GC para evitar las mismas complicaciones que la PCR.
Durante los ciclos de la polimerasa, los oligonucleótidos se hibridan con los fragmentos complementarios y luego se rellenan con la polimerasa. Cada ciclo aumenta así la longitud de varios fragmentos aleatoriamente dependiendo de qué oligonucleótidos se encuentran entre sí. Es fundamental que exista complementariedad entre todos los fragmentos de alguna manera o no se producirá una secuencia final completa ya que la polimerasa requiere un molde a seguir.
Después de esta fase de construcción inicial, se agregan cebadores adicionales que abarcan ambos extremos para realizar una reacción de PCR regular, amplificando la secuencia diana de todos los fragmentos incompletos más cortos. A continuación, se puede utilizar una purificación en gel para identificar y aislar la secuencia completa.
Reacción típica
Una reacción típica consiste en oligonucleótidos de ~ 50 pares de bases de largo, cada uno superpuesto por aproximadamente 20 pares de bases. La reacción con todos los oligonucleótidos se lleva a cabo durante ~ 30 ciclos seguidos de 23 ciclos adicionales con los cebadores finales.
Montaje de Gibson
Una modificación de este método, ensamblaje de Gibson , descrito por Gibson et al. permite el ensamblaje isotérmico de ADN en un solo paso con varios cientos de kb . Mediante el uso de exonucleasa T5 para "masticar" los extremos complementarios, se puede crear una superposición de aproximadamente 40 pb. La reacción tiene lugar a 50 ° C, temperatura en la que la exonucleasa T5 es inestable. Después de un breve intervalo de tiempo, se degrada, las superposiciones se pueden templar y ligar. El equipo IGEM de la Universidad de Cambridge hizo un video que describe el proceso. La clonación independiente de la ligadura (LIC) es una nueva variante del método para compilar varias piezas de ADN juntas y solo necesita la enzima exonucleasa para la reacción. Sin embargo, el método requiere la unión de un número par de piezas de ADN y la síntesis (generalmente mediada por PCR) de adaptadores adecuados. Por tanto, LIC no puede considerarse un método de subclonación "que no deja cicatrices".
Referencias
- Stemmer; et al. (1995). "Ensamblaje de un solo paso de un gen y plásmido completo de un gran número de oligodesoxirribonucleótidos". Gene . 164 (1): 49–53. doi : 10.1016 / 0378-1119 (95) 00511-4 . PMID 7590320 .
- Herrero; et al. (2003). "Generación de un genoma sintético por ensamblaje de genoma completo: bacteriófago [var phi] X174 a partir de oligonucleótidos sintéticos" . PNAS . 100 (26): 15440-15445. doi : 10.1073 / pnas.2237126100 . PMC 307586 . PMID 14657399 .
- ^ Stemmer; et al. (1995). "Ensamblaje de un solo paso de un gen y plásmido completo de un gran número de oligodesoxirribonucleótidos". Gene . 164 (1): 49–53. doi : 10.1016 / 0378-1119 (95) 00511-4 . PMID 7590320 .
- ^ Gibson DG, Young L, Chuang RY, Venter JC, Hutchison CA 3rd, Smith HO (2009). "Ensamblaje enzimático de moléculas de ADN de hasta varios cientos de kilobases". Métodos de la naturaleza . 6 (5): 343–345. doi : 10.1038 / nmeth.1318 . PMID 19363495 . S2CID 1351008 .
- ^ Vídeo de Gibson Assembly en YouTube