Rata knockout - Knockout rat
Una rata knock-out es una rata modificada genéticamente con un solo gen desactivado a través de una mutación dirigida ( captura de genes ) que se utiliza para la investigación académica y farmacéutica . Las ratas knockout pueden imitar enfermedades humanas y son herramientas importantes para estudiar la función genética ( genómica funcional ) y para el descubrimiento y desarrollo de fármacos . La producción de ratas knockout no fue económica o técnicamente viable hasta 2008.
La tecnología desarrollada a través de la financiación de los Institutos Nacionales de Salud (NIH) y el trabajo realizado por los miembros del Consorcio Knock Out Rat (KORC) llevó a métodos rentables para crear ratas knockout. La importancia de desarrollar la rata como una herramienta más versátil para la investigación en salud humana se evidencia en la inversión de $ 120 millones realizada por los NIH a través del Consorcio del Proyecto de Secuenciación del Genoma de Ratas, que resultó en la secuencia preliminar de una cepa de laboratorio de la rata marrón o noruega ( Rattus norvegicus ). Desarrollos adicionales con la tecnología de nucleasa de dedos de zinc en 2009 llevaron a la primera rata knockout con mutaciones dirigidas transmitidas por la línea germinal. SAGE Labs está comercializando modelos de enfermedad de rata knockout para Parkinson , Alzheimer , hipertensión y diabetes que utilizan tecnología de nucleasa de dedos de zinc.
Uso de investigación
Los ratones, las ratas y los humanos comparten todos los genes de los demás, excepto aproximadamente el 1%, lo que hace que los roedores sean buenos organismos modelo para estudiar la función genética humana. Tanto los ratones como las ratas son relativamente pequeños, fáciles de manipular, tienen un tiempo de generación corto y son genéticamente endogámicos. Si bien los ratones han demostrado ser un modelo útil de roedores y se han desarrollado técnicas para la alteración rutinaria de sus genes, en muchas circunstancias las ratas se consideran un animal de laboratorio superior para estudiar y modelar enfermedades humanas.
Las ratas son fisiológicamente más similares a los humanos que los ratones. Por ejemplo, las ratas tienen una frecuencia cardíaca más similar a la de los humanos, mientras que los ratones tienen una frecuencia cardíaca de cinco a diez veces más rápida. Se cree ampliamente que la rata es un modelo mejor que el ratón para enfermedades cardiovasculares humanas , diabetes, artritis y muchos trastornos autoinmunitarios , neurológicos , conductuales y de adicción. Además, los modelos de rata son superiores a los modelos de ratón para probar la farmacodinámica y la toxicidad de compuestos terapéuticos potenciales, en parte porque el número y el tipo de muchas de sus enzimas desintoxicantes son muy similares a los de los humanos. Su tamaño más grande hace que las ratas sean más propicias para el estudio por instrumentación y también facilita la manipulación, como la toma de muestras de sangre, la conducción nerviosa y la realización de cirugías.
Las técnicas para la manipulación genética están disponibles en el ratón, que se usa comúnmente para modelar enfermedades humanas. Aunque existen knockouts publicados para aproximadamente el 60% de los genes de ratón, una gran mayoría de enfermedades humanas comunes no tienen un modelo de ratón knockout . Los modelos de rata knockout son una alternativa a los ratones que pueden permitir la creación de nuevas alteraciones genéticas que no están disponibles en el ratón. Los modelos de ratas knockout también pueden complementar los modelos de ratones transgénicos existentes. La comparación de mutantes de ratón y rata puede facilitar la distinción entre fenotipos de mamíferos generales y específicos de roedores .
Desafíos de producción
Los modelos de rata se han utilizado para avanzar en muchas áreas de la investigación médica, incluidas enfermedades cardiovasculares, trastornos psiquiátricos (estudios de intervención conductual y adicción), regeneración neuronal , diabetes, trasplantes , trastornos autoinmunes ( artritis reumatoide ), cáncer y cicatrización de heridas y huesos. Si bien la finalización de la secuencia del genoma de la rata proporciona información muy clave, la forma en que estas enfermedades se relacionan con la función de los genes requiere un método eficaz para crear modelos de rata knockout en los que se manipulen secuencias genómicas específicas. La mayoría de las técnicas de manipulación genética, incluida la mutagénesis aleatoria con una trampa genética (basada en retroviral y no retroviral), knock-outs / knock-ins de genes y mutaciones condicionales, dependen del cultivo y manipulación del tallo embrionario (ES) células. Las células ES de rata se aislaron sólo recientemente y no se ha informado de ninguna demostración de modificación genética en ellas. En consecuencia, muchas técnicas de manipulación genética ampliamente utilizadas en el ratón no son posibles en la rata.
Primeros métodos
Hasta el desarrollo comercial de la tecnología de ADN móvil en 2007 y la tecnología de nucleasa con dedos de zinc en 2009, solo había dos tecnologías que podían usarse para producir modelos de ratas de enfermedades humanas: la clonación y la mutagénesis química utilizando N-etil-N-nitrosourea ( ENU ). Aunque la clonación por transferencia nuclear de células somáticas (SCNT) podría teóricamente usarse para crear ratas con mutaciones específicas mediante la mutación de células somáticas y luego usar estas células para SCNT, este enfoque no se ha utilizado con éxito para crear ratas knockout. Un problema con esta estrategia es que SCNT es extremadamente ineficiente. El primer intento publicado tuvo una tasa de éxito de menos del 1%. Alternativamente, la mutagénesis de ENU es una estrategia común de eliminación de genes de mutagénesis aleatoria en el ratón que también puede usarse en la rata. La mutagénesis de ENU implica el uso de una sustancia química, N-etil-N-nitrosourea (ENU), para crear cambios de base única en el genoma. ENU transfiere su grupo etilo a radicales de oxígeno o nitrógeno en el ADN, lo que resulta en un emparejamiento incorrecto y sustitución de pares de bases. Se pueden producir animales mutantes inyectando ENU a un ratón macho y reproduciéndolo con una hembra de tipo salvaje para producir descendencia mutante. La mutagénesis ENU crea una alta frecuencia de mutaciones aleatorias, con aproximadamente un cambio de par de bases en cualquier gen dado por cada 200-700 gametos. A pesar de su alta mutagenicidad, la penetración física de ENU es limitada y solo unos 500 genes mutan para cada macho y un número muy pequeño de las mutaciones totales tienen un fenotipo observable. Por lo general, es necesario crear miles de mutaciones en un solo animal para generar un fenotipo nuevo.
A pesar de las recientes mejoras en la tecnología ENU, el mapeo de mutaciones responsables de un fenotipo en particular suele ser difícil y requiere mucho tiempo. Las mutaciones neutrales deben separarse de las mutaciones causales mediante la reproducción extensiva. Los métodos ENU y de clonación son simplemente ineficaces para crear y mapear knockouts de genes en ratas para la creación de nuevos modelos de enfermedades humanas. Hasta 2007, el proyecto de mutagénesis de ENU en ratas más grande hasta la fecha dirigido por el Medical College of Wisconsin pudo producir solo 9 líneas de ratas knockout en un período de cinco años a un costo promedio de $ 200,000 por línea knockout. Aunque algunas empresas todavía están aplicando esta estrategia, el Medical College of Wisconsin ha cambiado a un método más eficiente y comercialmente viable que utiliza ADN móvil y tecnología CompoZr ZFN.
Tecnología de nucleasa de dedos de zinc y TALE
Las nucleasas de dedos de zinc (ZFN) y las nucleasas efectoras de tipo activador de la transcripción (TALEN) son proteínas de unión al ADN diseñadas que facilitan la edición dirigida del genoma mediante la creación de rupturas de doble cadena en el ADN en ubicaciones especificadas por el usuario. Las roturas de doble hebra son importantes para la mutagénesis de un sitio específico porque estimulan los procesos naturales de reparación del ADN de la célula, a saber, la recombinación homóloga y la unión de extremos no homólogos. Cuando la célula utiliza la vía de unión del extremo no homóloga para reparar la rotura de la doble hebra, la inexactitud inherente de la reparación a menudo genera mutaciones dirigidas con precisión. Esto da como resultado embriones con eliminación genética dirigida. Las técnicas de microinyección estándar permiten que esta tecnología produzca ratas knockout en 4-6 meses. Una ventaja importante de la desactivación génica mediada por ZFN y TALEN en relación con el uso de ADN móvil es que un gen particular puede dirigirse de forma única y específica para la desactivación. Por el contrario, los knockouts realizados con tecnología de ADN móvil son aleatorios y, por lo tanto, es poco probable que apunten al gen de interés.
Tecnología de ADN móvil
La tecnología de ADN móvil (gen saltarín) utiliza retrotransposones y transposones para la producción de modelos de rata knockout. Esta tecnología de plataforma cumple con todos los criterios para un enfoque exitoso de eliminación de genes en mamíferos al permitir la mutagénesis aleatoria directamente en las células germinales ( espermatozoides y ovocitos ) de organismos modelo de mamíferos, incluidas las ratas. Con esta tecnología, los genes se alteran por completo y de manera estable, se eliminan con una frecuencia alta y se alteran aleatoriamente en todo el genoma. La ubicación genómica de las mutaciones se puede mapear fácilmente, creando una biblioteca de ratas knockout para su uso posterior. Una vez que se crean las mutaciones knockout aleatorias, se pueden crear mutaciones más refinadas, como mutaciones condicionales, mediante la reproducción de líneas knockout con líneas de rata que expresan la recombinasa CRE de una manera específica de tejido. Los knock-ins se pueden producir mediante intercambio de casetes mediado por recombinación.
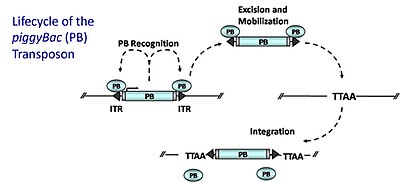
transposones de ADN piggyBac (PB)
Los transposones de ADN piggyBac (PB) se movilizan a través de un mecanismo de "cortar y pegar" mediante el cual una enzima transposasa (PB transposasa), codificada por el transposón mismo, escinde y reintegra el transposón en otros sitios dentro del genoma. La transposasa PB reconoce específicamente las repeticiones terminales invertidas (ITR) de PB que flanquean el transposón; se une a estas secuencias y cataliza la escisión del transposón. Luego, PB se integra en sitios TTAA en todo el genoma, de una manera relativamente aleatoria. Para la creación de mutaciones de trampa de genes (o adaptadas para generar animales transgénicos), la transposasa se suministra en trans en un plásmido y se cotransfecta con un plásmido que contiene el transposón donante, un transposón recombinante que comprende una trampa de genes flanqueada por los sitios de unión para la transposasa (ITR). La transposasa catalizará la escisión del transposón del plásmido y la posterior integración en el genoma. La integración dentro de una región codificante capturará los elementos necesarios para la expresión de la trampa de genes. PB posee varias propiedades ideales: (1) se inserta preferentemente dentro de los genes (50 a 67% de las inserciones golpean genes) (2) no exhibe saltos locales (cobertura genómica generalizada) (3) no es sensible a la inhibición de la sobreproducción en cuyos niveles elevados de transposasa provocan una disminución de la transposición 4) se escinde limpiamente del sitio donante, sin dejar “huella”, a diferencia de la Bella Durmiente.
Transposones de la bella durmiente (SB)
El transposón de la bella durmiente (SB) es un derivado de la superfamilia Tc1 / mariner de transposones de ADN que prevalecen entre los genomas de vertebrados e invertebrados. Sin embargo, los transposones de ADN endógenos de esta familia son completamente inactivos en los genomas de vertebrados. Un transposón activo Tc1 / mariner, sintetizado a partir de la alineación de transposones inactivos de la subfamilia de elementos de los salmónidos, fue "despertado" para formar el transposón llamado La Bella Durmiente. SB, al igual que otros transposones de ADN, se moviliza a través de un mecanismo de cortar y pegar mediante el cual una enzima transposasa, codificada por el propio transposón, escinde y reintegra el transposón en otros sitios dentro del genoma. La proteína SB de 340 aminoácidos reconoce las repeticiones terminales invertidas (ITR) que flanquean el transposón; se une a estas secuencias y cataliza la escisión del transposón. SB luego se integra en sitios aleatorios dentro del genoma, aunque algunos estudios informan preferencias muy leves por las unidades transcripcionales. También existe un requisito simple de un dinucleótido TA en el sitio objetivo, como todos los transposones Tc1 / mariner.
El transposón SB es una herramienta poderosa para la mutagénesis de inserción en muchas especies de vertebrados. Recientemente ha mostrado una utilidad especial para la mutagénesis de la línea germinal tanto en ratones como en ratas. Hay varias ventajas que hacen de SB un mutágeno altamente atractivo orientado al descubrimiento de genes: 1) tiene poco sesgo para insertarse dentro de regiones genómicas particulares o dentro de secuencias de reconocimiento específicas, 2) las inserciones de novo del transposón proporcionan un marcador de secuencia "marcado" para identificación rápida de la mutación específica mediante métodos simples de clonación por PCR, 3) la mutagénesis de inserción de SB in vivo permite generar múltiples mutaciones rápida y fácilmente en un solo animal y en un solo tejido, como un pólipo adenomatoso.
Retrotransposones LINE1 (L1)
Los transposones y retrotransposones son herramientas valiosas para el descubrimiento de genes sin sesgos como piezas móviles de ADN que se utilizan para la alteración de genes. Los retrotransposones, como los LINE (elementos nucleares intercalados largos), se movilizan mediante un mecanismo de "copiar y pegar" y son abundantes en muchas especies eucariotas. Varios retrotransposones L1 han permanecido activos en ratones y humanos. Los L1 contienen un pequeño promotor interno dentro de una región no traducida 5 'para impulsar la expresión, dos marcos de lectura abiertos (ORF) y una región no traducida 3' que contiene secuencias para la poliadenilación. Los dos ORF codifican proteínas necesarias para la retrotransposición autónoma; ORF1 codifica una proteína de unión a ARN, mientras que ORF2 codifica una proteína que contiene actividad de endonucleasa (EN) y transcriptasa inversa (RT), que corta un sitio en el ADN y luego produce una copia a través de RT. Estas proteínas exhiben una abrumadora especificidad para unirse y actuar sobre la transcripción que las codifica, lo que permite una movilización casi exclusiva del ARN L1 parental. Usando la actividad de RT de la proteína ORF2, el ARN de L1 transcrito se copia en el ADN mediante un proceso denominado transcripción inversa cebada por diana (TPRT) y se integra en el genoma. La integración ocurre con poco sesgo para cualquier región genómica particular, lo que requiere una secuencia de consenso simple, 5'TTTT'A-3 '(junto con variaciones menores de esta secuencia). Las secuencias L1 integradas a menudo se truncan en el extremo 5 ', con un tamaño total medio de 1 Kb, y muchas contienen solo secuencias terminales 3'.
La naturaleza de la retrotransposición confiere al L1 algunas ventajas únicas; Los retrotransposones L1 tienen un suministro esencialmente ilimitado del mutágeno de inserción ya que se transcribe continuamente a partir de un promotor, lo que sería útil para aplicaciones en las que se necesita un gran número de mutaciones en una sola célula. Los elementos L1 también demuestran una cobertura genómica generalizada, con una distribución de inserciones en gran parte aleatoria. Las inserciones de L1 en los sitios genómicos también son irreversibles y, por lo tanto, cualquier evento mutagénico causado por una inserción de L1 está "marcado" por secuencias de L1.
Ver también
Referencias
enlaces externos
- NIH Genoma y genética de ratas
- Los científicos comparan el genoma de la rata con el humano, el ratón
- Sitio web del Consorcio Knock Out Rat (KORC)
- Sitio web de los Institutos Nacionales de Salud
- Transposagen: productores de modelos de ratas Knockout
- Knockout Rats - Horizon Discovery (anteriormente SAGE Labs)