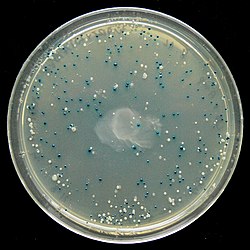
Pantalla azul-blanca - Blue–white screen
La pantalla azul-blanca es una técnica de detección que permite la detección rápida y conveniente de bacterias recombinantes en experimentos de clonación molecular basados en vectores . Este método de cribado se realiza habitualmente utilizando una cepa bacteriana adecuada , pero también se pueden utilizar otros organismos como la levadura. El ADN de interés se liga a un vector . A continuación, el vector se inserta en una célula huésped competente viable para la transformación, que luego se hace crecer en presencia de X-gal . Las células transformadas con vectores que contienen ADN recombinante producirán colonias blancas; las células transformadas con plásmidos no recombinantes (es decir, solo el vector) crecen en colonias azules.
Fondo
La clonación molecular es uno de los procedimientos más utilizados en biología molecular . Puede insertarse un gen de interés en un vector plasmídico mediante ligación , y luego el plásmido se transforma en células de Escherichia coli . Sin embargo, no todos los plásmidos transformados en células pueden contener el inserto de gen deseado, y comprobar la presencia del inserto en cada colonia individual lleva mucho tiempo. Por lo tanto, un método para la detección del inserto sería útil para hacer que este procedimiento requiera menos tiempo y trabajo. Uno de los primeros métodos desarrollados para la detección de insertos es el cribado azul-blanco que permite la identificación de productos exitosos de reacciones de clonación a través del color de la colonia bacteriana .
El método se basa en el principio de complementación α del gen de la β-galactosidasa . Este fenómeno de α-complementación se demostró por primera vez en el trabajo realizado por Agnes Ullmann en el laboratorio de François Jacob y Jacques Monod , donde se demostró que la función de una β-galactosidasa mutante inactiva con secuencia eliminada se rescataba mediante un fragmento de β-galactosidasa. en el que esa misma secuencia, el péptido donante α, todavía está intacta. Langley y col. mostró que la β-galactosidasa mutante no funcional carecía en parte de su extremo N-terminal con sus residuos 11-41 suprimidos, pero puede complementarse con un péptido formado por los residuos 3-90 de β-galactosidasa. El fago filamentoso M13 que contiene la secuencia que codifica los primeros 145 aminoácidos fue posteriormente construido por Messing et al. , y la complementación α mediante el uso de un vector se demostró mediante la formación de placas azules cuando las células que contenían la proteína inactiva fueron infectadas por el fago y luego se cultivaron en placas que contenían X-gal.
La serie pUC de vectores de clonación de plásmidos de Vieira y Messing se desarrolló a partir del sistema M13 y fueron los primeros plásmidos construidos para aprovechar este método de selección. En este método, el ADN ligado al plásmido interrumpe el péptido α y, por lo tanto, el proceso de complementación, y no se puede formar β-galactosidasa funcional. Por tanto, las células transformadas con plásmido que contiene un inserto forman colonias blancas, mientras que las células transformadas con plásmido sin inserto forman colonias azules; El resultado de una ligadura satisfactoria puede identificarse fácilmente por la coloración blanca de las células formadas a partir de las azules fallidas.
Mecanismo molecular
La β-galactosidasa es una proteína codificada por el gen lacZ del operón lac y existe como homotetrámero en su estado activo. Sin embargo, una β-galactosidasa mutante derivada de la cepa M15 de E. coli tiene sus residuos N-terminales 11-41 eliminados y este mutante, el péptido, no puede formar un tetrámero y está inactivo. Sin embargo, esta forma mutante de proteína puede volver completamente a su estado tetramérico activo en presencia de un fragmento N-terminal de la proteína, el péptido α. El rescate de la función de la β-galactosidasa mutante por el péptido α se denomina complementación α.
En este método de cribado, la cepa de E. coli del hospedador porta el mutante de deleción lacZ ( lacZΔM15 ) que contiene el péptido ω, mientras que los plásmidos usados llevan la secuencia lacZα que codifica los primeros 59 residuos de la β-galactosidasa, el péptido α . Ninguno de los dos es funcional por sí mismo. Sin embargo, cuando los dos péptidos se expresan juntos, como cuando un plásmido que contiene el lacZα secuencia se transforma en un lacZΔM15 células, forman un funcional β-galactosidasa enzima.
El método de tramado azul / blanco funciona interrumpiendo este proceso de complementación α. El plásmido lleva dentro de la secuencia lacZα un sitio interno de clonación múltiple (MCS). Este MCS dentro de la secuencia lacZα puede cortarse mediante enzimas de restricción de modo que el ADN extraño pueda insertarse dentro del gen lacZ α, interrumpiendo así el gen que produce el péptido α. En consecuencia, en las células que contienen el plásmido con un inserto, no se puede formar β-galactosidasa funcional.
La presencia de una β-galactosidasa activa puede detectarse mediante X-gal , un análogo incoloro de la lactosa que puede ser escindido por β-galactosidasa para formar 5-bromo-4-cloro-indoxil, que luego se dimeriza y oxida espontáneamente para formar un Pigmento insoluble azul brillante 5,5'-dibromo-4,4'-dicloro-índigo. Esto da como resultado un color azul característico en las células que contienen una β-galactosidasa funcional. Por lo tanto, las colonias azules muestran que pueden contener un vector con una lacZα ininterrumpida (por lo tanto, sin inserto), mientras que las colonias blancas, donde X-gal no está hidrolizado, indican la presencia de un inserto en lacZα que interrumpe la formación de una β-galactosidasa activa. .
Los clones recombinantes pueden analizarse adicionalmente aislando y purificando pequeñas cantidades de ADN plasmídico de las colonias transformadas y pueden usarse enzimas de restricción para cortar el clon y determinar si tiene el fragmento de interés. Si es necesario secuenciar el ADN, los plásmidos de las colonias deberán aislarse en un punto, ya sea para cortar utilizando enzimas de restricción o realizando otros ensayos.
Consideraciones prácticas
El tipo correcto de vector y las células competentes son consideraciones importantes al planificar una pantalla azul blanca. El plásmido debe contener lacZ α, y ejemplos de tales plásmidos son pUC19 y pBluescript . La célula de E. coli debe contener el gen lacZ mutante con secuencia delecionada (es decir, lacZΔM15 ), y algunas de las células comúnmente utilizadas con dicho genotipo son JM109, DH5α y XL1-Blue. También debe entenderse que el operón lac se ve afectado por la presencia de glucosa. La proteína EIIA Glc , que participa en la importación de glucosa, cierra la lactosa permeasa cuando la glucosa se transporta al interior de la célula. Por tanto, los medios utilizados en la placa de agar no deben incluir glucosa.
X-gal es sensible a la luz y, por lo tanto, su solución y las placas que contienen X-gal deben almacenarse en la oscuridad. El isopropil β-D-1-tiogalactopiranósido (IPTG), que funciona como inductor del operón lac , puede usarse en los medios para mejorar la expresión de LacZ.
X-gal es un material caro, por lo que se han desarrollado otros métodos para detectar bacterias. GFP se ha desarrollado como una alternativa para ayudar a detectar bacterias. El concepto es similar a la complementación α en la que un inserto de ADN puede interrumpir la secuencia codificante dentro de un vector y, por lo tanto, interrumpir la producción de GFP dando como resultado bacterias no fluorescentes. Las bacterias que tienen vectores recombinantes (vector + inserto), serán blancas y no expresarán la proteína GFP, mientras que las no recombinantes (vector), emitirán fluorescencia bajo luz ultravioleta. La GFP en general se ha utilizado como un gen informador en el que los individuos pueden determinar definitivamente si un clon es portador de un gen que los investigadores están analizando. En ocasiones, el medio en el que crecen las colonias puede influir en la pantalla e introducir resultados falsos positivos. X-gal en el medio puede degradarse ocasionalmente para producir un color azul o GFP puede perder su fluorescencia debido al medio y puede afectar la capacidad de los investigadores para determinar las colonias con el recombinante deseado y las que no lo poseen.
Inconvenientes
Algunas colonias blancas pueden no contener el plásmido recombinante deseado por varias razones. El ADN ligado puede no ser el correcto o no estar ligado apropiadamente, y es posible que algún vector linealizado se transforme, sus extremos "reparados" y ligados entre sí de manera que no se produzca LacZα y no se formen colonias azules. La mutación también puede conducir a que el fragmento α no se exprese. Una colonia sin ningún vector también aparecerá de color blanco y, a veces, puede aparecer como colonias satélite después de que se haya agotado el antibiótico utilizado. También es posible que las colonias azules contengan el inserto. Esto ocurre cuando el inserto está "en marco" con el gen LacZα y no hay un codón STOP en el inserto. Esto puede conducir a la expresión de una proteína de fusión que tiene un LacZα funcional si su estructura no se altera. La construcción recombinante correcta a veces puede dar colonias de color azul más claro que pueden complicar su identificación.