Inhibidor de proteasa (biología) - Protease inhibitor (biology)
En biología y bioquímica , los inhibidores de proteasas , o antiproteasas , son moléculas que inhiben la función de las proteasas ( enzimas que ayudan a descomponer las proteínas ). Muchos inhibidores de proteasa de origen natural son proteínas .
En medicina , el inhibidor de la proteasa a menudo se usa indistintamente con la alfa 1-antitripsina (A1AT, que se abrevia PI por esta razón). La A1AT es, de hecho, el inhibidor de proteasa implicado con mayor frecuencia en la enfermedad, es decir, en la deficiencia de alfa-1 antitripsina .
Clasificación
Los inhibidores de proteasa pueden clasificarse por el tipo de proteasa que inhiben o por su mecanismo de acción. En 2004, Rawlings y sus colegas introdujeron una clasificación de inhibidores de proteasa basada en similitudes detectables a nivel de secuencia de aminoácidos. Esta clasificación identificó inicialmente 48 familias de inhibidores que podrían agruparse en 26 superfamilias (o clanes) relacionados por su estructura. Según la base de datos MEROPS, ahora hay 81 familias de inhibidores. Estas familias se nombran con una I seguida de un número, por ejemplo, I14 contiene inhibidores similares a la hirudina .
Por proteasa
Las clases de proteasas son:
- Inhibidores de la proteasa aspártica
- Inhibidores de la cisteína proteasa
- Inhibidores de metaloproteasas
- Inhibidores de la serina proteasa
- Inhibidores de treonina proteasa
- Inhibidores de tripsina
Por mecanismo
Las clases de mecanismos de acción inhibidores son:
- Inhibidor de suicidio
- Inhibidor del estado de transición
- Inhibidor de proteasa proteasa (ver serpinas )
- Agentes quelantes
Familias
Inhibidor I4
Esta es una familia de inhibidores suicidas de proteasas llamados serpinas . Contiene inhibidores de múltiples familias de proteasas de cisteína y serina. Su mecanismo de acción se basa en sufrir un gran cambio conformacional que inactiva la tríada catalítica de su objetivo .
Inhibidor I9
| Inhibidor de peptidasa I9 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
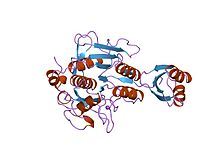 subtilisina bpn 'prosegment (77 residuos) complejado con una subtilisina bpn mutante' (266 residuos). pH cristalino 4,6. temperatura de cristalización 20 c temperatura de difracción-160 c
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I9 | ||||||||
| Pfam | PF05922 | ||||||||
| InterPro | IPR010259 | ||||||||
| MEROPS | I9 | ||||||||
| SCOP2 | 1gns / SCOPe / SUPFAM | ||||||||
| |||||||||
Los inhibidores de propéptidos de proteinasa (a veces denominados péptidos de activación) son responsables de la modulación del plegamiento y la actividad de la proenzima peptidasa o zimógeno . El pro-segmento se acopla a la enzima, protegiendo el sitio de unión del sustrato , promoviendo así la inhibición de la enzima. Varios de estos propéptidos comparten una topología similar, a pesar de las identidades de secuencia a menudo bajas . La región del propéptido tiene un pliegue de sándwich abierto antiparalelo-alfa / antiparalelo-beta, con dos hélices alfa y cuatro hebras beta con una topología (beta / alfa / beta) x2. La familia I9 del inhibidor de la peptidasa contiene el dominio propéptido en el extremo N-terminal de las peptidasas que pertenecen a la familia S8A de MEROPS, subtilisinas . El propéptido se elimina mediante escisión proteolítica; eliminación activando la enzima.
Inhibidor I10
| Inhibidores de la endopeptidasa de serina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 estructura en solución de marinostatina, un inhibidor de proteasa, que contiene dos enlaces éster
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I10 | ||||||||
| Pfam | PF12559 | ||||||||
| InterPro | IPR022217 | ||||||||
| |||||||||
Esta familia incluye tanto microviridinas como marinostatinas. Parece probable que en ambos casos sea el extremo C el que se convierta en el inhibidor activo después de modificaciones postraduccionales del prepéptido de longitud completa. Son los enlaces éster dentro de la región clave de 12 residuos los que circularizan la molécula dándole su conformación inhibidora .
Inhibidor I24
| Inhibidor de la peptidasa PinA | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Inhibidor_I24 | ||||||||
| Pfam | PF10465 | ||||||||
| InterPro | IPR019506 | ||||||||
| MEROPS | I24 | ||||||||
| |||||||||
Esta familia incluye PinA, que inhibe la endopeptidasa La. Se une al homotetramero de La pero no interfiere con el sitio de unión de ATP o el sitio activo de La.
Inhibidor I29
| Dominio inhibidor del propéptido de catepsina (I29) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
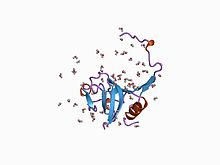 estructura cristalina de una proforma de cisteína proteasa
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I29 | ||||||||
| Pfam | PF08246 | ||||||||
| InterPro | IPR013201 | ||||||||
| |||||||||
El dominio inhibidor I29 , que pertenece a la familia I29 del inhibidor de la peptidasa MEROPS, se encuentra en el extremo N de una variedad de precursores de la peptidasa que pertenecen a la subfamilia C1A de la peptidasa MEROPS; estos incluyen catepsina L, papaína y procaricaína. Forma un dominio de hélice alfa que atraviesa el sitio de unión al sustrato, impidiendo el acceso. La eliminación de esta región por escisión proteolítica da como resultado la activación de la enzima . Este dominio también se encuentra, en una o más copias, en una variedad de inhibidores de la cisteína peptidasa como la salarina.
Inhibidor I34
| Inhibidor de la saccharopepsina I34 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 la estructura de la proteinasa a complejada con un inhibidor mutante ia3
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I34 | ||||||||
| Pfam | PF10466 | ||||||||
| InterPro | IPR019507 | ||||||||
| MEROPS | I34 | ||||||||
| |||||||||
El inhibidor de la sacaropepsina I34 es muy específico para la saccharopepsina peptidasa aspártica. En ausencia de sacaropepsina, está en gran parte desestructurado, pero en su presencia, el inhibidor sufre un cambio conformacional que forma una hélice alfa casi perfecta de Asn 2 a Met 32 en la hendidura del sitio activo de la peptidasa.
Inhibidor I36
| Familia de inhibidores de peptidasa I36 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
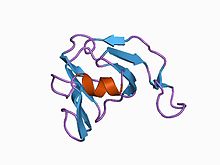 la estructura 3d del inhibidor de metaloproteinasas de streptomyces, smpi, aislado de streptomyces nigrescens tk-23, nmr, estructura promedio minimizada
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I36 | ||||||||
| Pfam | PF03995 | ||||||||
| Clan pfam | CL0333 | ||||||||
| InterPro | IPR007141 | ||||||||
| MEROPS | I36 | ||||||||
| SCOP2 | 1bhu / SCOPe / SUPFAM | ||||||||
| |||||||||
El dominio I36 de la familia de inhibidores de peptidasas solo se encuentra en un pequeño número de proteínas restringidas a la especie Streptomyces . Todos tienen cuatro cisteínas conservadas que probablemente forman dos enlaces disulfuro . Una de estas proteínas de Streptomyces nigrescens es el inhibidor de metaloproteinasas SMPI bien caracterizado .
Se ha determinado la estructura de SMPI. Tiene 102 residuos de aminoácidos con dos puentes disulfuro e inhibe específicamente las metaloproteinasas como la termolisina , que pertenece a la familia de peptidasas MEROPS M4. SMPI se compone de dos hojas beta , cada una de las cuales consta de cuatro hebras beta antiparalelas . La estructura se puede considerar como dos motivos de clave griega con simetría interna doble, un barril beta de clave griega . Una característica estructural única que se encuentra en SMPI es su extensión entre la primera y la segunda hebras del segundo motivo clave griega que se sabe que está involucrado en la actividad inhibidora de SMPI. En ausencia de similitud de secuencia, la estructura de SMPI muestra una clara similitud con ambos dominios de las cristalinas del cristalino del ojo , ambos dominios de la proteína S sensora de calcio, así como la toxina asesina de levaduras de dominio único . Se pensaba que la estructura de la toxina asesina de levaduras era un precursor de las proteínas beta gamma-cristalinas de dos dominios, debido a su similitud estructural con cada dominio de las beta gamma-cristalinas. Por tanto, SMPI proporciona otro ejemplo de una estructura de proteína de dominio único que corresponde al pliegue ancestral a partir del cual se cree que han evolucionado las proteínas de dos dominios en la superfamilia de beta gamma-cristalina .
Inhibidor I42
| Inhibidor de la peptidasa de la familia de la chagasina I42 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 estructura de la solución del inhibidor de la cisteína proteasa tripanosoma cruzi chagasina
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I42 | ||||||||
| Pfam | PF09394 | ||||||||
| InterPro | IPR018990 | ||||||||
| MEROPS | I42 | ||||||||
| |||||||||
La familia de inhibidores I42 incluye la chagasina, un inhibidor reversible de las cisteína proteasas similares a la papaína . La chagasina tiene una estructura de barril beta , que es una variante única del pliegue de inmunoglobulina con homología con la CD8 alfa humana .
Inhibidor I48
| Inhibidor de peptidasa clitocipina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Inhibidor_I48 | ||||||||
| Pfam | PF10467 | ||||||||
| InterPro | IPR019508 | ||||||||
| MEROPS | I48 | ||||||||
| |||||||||
La familia de inhibidores I48 incluye la clitocipina, que se une e inhibe las cisteína proteinasas. No tiene ninguna similitud con cualquier otro inhibidores de proteinasas de cisteína conocida pero tiene cierta similitud con una lectina -como familia de proteínas de hongos .
Inhibidor I53
| Inhibidor de trombina Madanin | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Inhibidor_I53 | ||||||||
| Pfam | PF11714 | ||||||||
| InterPro | IPR021716 | ||||||||
| MEROPS | I53 | ||||||||
| |||||||||
Los miembros de esta familia son las proteínas inhibidoras de la peptidasa madanina . Estas proteínas se aislaron de la saliva de las garrapatas .
Inhibidor I67
| Inhibidor de bromelina VI | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 estructura de RMN del inhibidor de bromelina vi del tallo de la piña
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I67 | ||||||||
| Pfam | PF11405 | ||||||||
| InterPro | IPR022713 | ||||||||
| MEROPS | I67 | ||||||||
| |||||||||
Bromelina inhibidor VI, en la familia de inhibidores I67, es una de cadena doble inhibidor que consiste en un 11 residuos y un 41-residuo cadena .
Inhibidor I68
| Inhibidor de carboxipeptidasa I68 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 estructura cristalina del inhibidor de la carboxipeptidasa de garrapatas en un complejo con la carboxipeptidasa b humana
| |||||||||
| Identificadores | |||||||||
| Símbolo | Inhibidor_I68 | ||||||||
| Pfam | PF10468 | ||||||||
| InterPro | IPR019509 | ||||||||
| MEROPS | I68 | ||||||||
| |||||||||
La familia de inhibidores de carboxipeptidasa I68 representa una familia de inhibidores de carboxipetidasa de garrapatas.
Inhibidor I78
| Familia de inhibidores de peptidasa I78 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Inhibitor_I78 | ||||||||
| Pfam | PF11720 | ||||||||
| Clan pfam | CL0367 | ||||||||
| InterPro | IPR021719 | ||||||||
| MEROPS | I78 | ||||||||
| |||||||||
La familia de inhibidores de peptidasa I78 incluye el inhibidor de elastasa de Aspergillus .
Compuestos
- Aprotinina
- Bestatina
- Inhibidor de calpaína I y II
- Quimostatina
- S-64
- Leupeptina ( N -acetil- L -leucil- L -leucil- L -argininal)
- alfa-2-macroglobulina
- Pefabloc SC
- Pepstatin
- PMSF (fluoruro de fenilmetanosulfonilo)
- TLCK
- Inhibidores de tripsina
